自然杂志上发表了一篇中国科研人员的论文《细菌细胞的混合培养可实现经济的DNA大规模存储》。
近日,自然杂志上发表了一篇中国科研人员的论文《细菌细胞的混合培养可实现经济的DNA大规模存储》,该文由天津大学化学工程与技术学院,天津生物系统工程教育部重点实验室,闵浩,乔红艳,高彦敏,王兆冠,王新冠,乔新等联合撰写。
文章摘要:
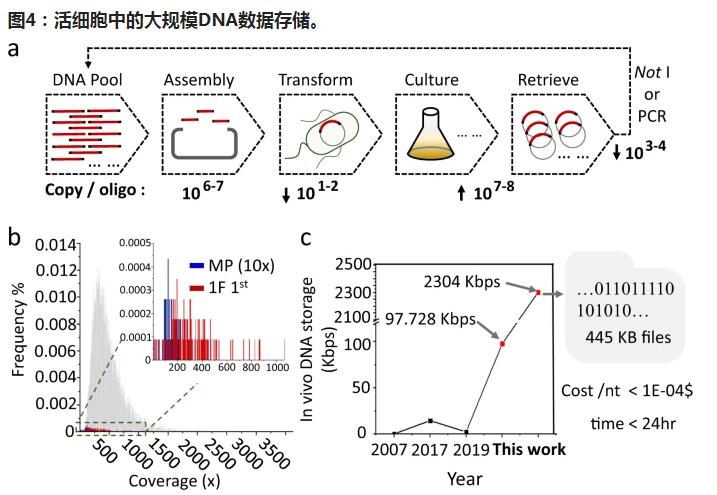
DNA成为海量数据存储的一种新型潜在材料,为廉价地解决大数据存储问题提供了可能性。较大的寡核苷酸池显示了在试管中进行大规模数据存储的巨大潜力,同时,活细胞在信息复制中具有很高的保真度。在这里,我们显示了携带大寡聚体的细菌细胞的混合培养物,该大寡聚体组装在高拷贝数质粒中,被作为稳定的材料用于大规模数据存储。通过深入的生物信息学分析探索了基本原理。尽管同源装配显示出序列背景依赖性偏差,但是混合培养物中的大寡核苷酸库在多个连续传代中是恒定的。最终,将超过2万个Kbps的10,000个不同的寡核苷酸编码为445 KB数字数据,并将其存储在单元中。
在这里,我们证明了带有大量寡核苷酸池的细菌细胞的混合培养物是一种经济且可持续的材料,用于稳定的信息存储,能够从高通量芯片合成中存储跨越数百个核苷酸的DNA寡核苷酸。BASIC代码系统是以前在我们实验室中开发的一种DNA介导的分布式信息存储系统,用于将数字二进制信息转换为核苷酸基序列,在软件级别的编码冗余度为1.56%,可以忍受少数寡核苷酸的物理缺失。通过突破极限,储存了由509和11520个不同的寡核苷酸组成的寡核苷酸池,这些细菌在细菌细胞的混合培养物中产生了最大的报告种群。为了覆盖非常少的寡头种群,我们以多余的方式组装它们,然后将它们以混合培养的形式保存在固体平板或液体培养基中。此外,使用专门开发的深度生物信息学分析工具探索了数据存储单元制造的基本原理。
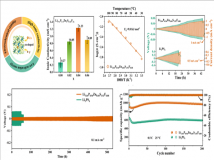
结果表明,寡核苷酸同源装配对序列上下文具有相对较高的偏差,并且寡核苷酸拷贝数分布随着装配中片段数目的增加而越来越不对称。然而,在组装和转化后,我们发现大量的寡聚核苷酸在混合培养中保持稳定。结果表明,寡核苷酸同源装配对序列上下文具有相对较高的偏差,并且寡核苷酸拷贝数分布随着装配中片段数目的增加而越来越不对称。然而,在组装和转化后,我们发现大量的寡聚核苷酸在混合培养中保持稳定。结果表明,寡核苷酸同源装配对序列上下文具有相对较高的偏差,并且寡核苷酸拷贝数分布随着装配中片段数目的增加而越来越不对称。然而,在组装和转化后,我们发现大量的寡聚核苷酸在混合培养中保持稳定。大肠杆菌细胞甚至经过多次传代,并保持了数字数据的质量,可以完美地进行信息解码。
最后,可以证明,这种基于细菌细胞混合培养的简单材料以快速,经济的方式实现了以445 KB的数字文件在总共2304 Kbps的合成DNA中的体内存储。据我们所知,这是迄今为止所报道的活细胞中规模最大的档案数据存储,这为以经济有效的方式利用活细胞的体外合成能力和生物能力两者进行生物数据存储铺平了道路。对于大规模开发实用的冷数据存储至关重要。
菌株和培养条件
使用具有电感受态能力的大肠杆菌DH10β进行克隆,并购自Biomed Co.,Ltd.(中国北京)。氨苄西林的抗生素使用量为100 mg / mL。除非另有说明,否则将细胞在Luria-Bertani(LB)肉汤中以220 rpm摇动培养,或在LB琼脂平板上于37°C培养。
图书馆建设
为了组装509个序列,合成了寡核苷酸库,并且冻干的库由192个nt的11,776个寡核苷酸(由Twist Bioscience合成)组成,每个寡核苷酸中包括152nt的有效载荷。将池重悬于1x TE缓冲液中,终浓度为2 ng /μL。其中一个文件509寡核苷酸的侧面是预混合引物F02 / R02的结合位点。PCR使用Q5®高保真DNA聚合酶(NEB#M0491)和引物F01-F04 / R01-F04(10 ng寡核苷酸,2.5μL每种引物混合物(100 mM),0.5μLQ5高保真DNA聚合酶,在50μL反应中加入4μL2.5 mM dNTP)。热循环条件如下:在98℃下5分钟;在室温下5分钟。10个周期:98°C下10 s,56°C下30 s,72°C下30 s,然后在72°C下延长5分钟。然后使用Plus DNA Clean / Extraction Kit(GMbiolab Co,Ltd.2 O.该库被认为是主池并在2%琼脂糖凝胶,以验证正确的大小运行。为了装配11520个序列,合成的DNA库由200个核苷酸的11520个寡核苷酸组成(由Twist Bioscience合成),其中包括155个核苷酸的有效负载,两侧是引物F1 / R1的结合位点(补充图 3)。将冻干的合并液在1x TE缓冲液中重新水化,并使用上述方案扩增文件。
引物同源臂设计的计算策略
引物包括三个部分,一个是用于吉布森装配的同源臂(同源臂),另一个是核酸内切酶识别位点的Not I,最后一个是地址序列。实施引物设计算法,以编码使用PCR生产装配片段的规则。引物同源臂是使用NUPACK(http://www.nupack.org)设计的。鉴定出满足以下规则的用于同源臂设计的引物组:(1)同源臂长度25nt;(2)鸟嘌呤-胞嘧啶(GC)含量为40-60%;(3)各同源臂之间无相互作用。通过所有上述测试的选定引物作为正确对提供,可产生定义大小的乘积(补充图 1)。和2)。
组装实验
对于主链制备,将pUC19质粒用作PCR的模板。使用最大DNA聚合酶(Takara#R045Q)和引物组PCR-vactor-F / R进行PCR。热循环扩增了30个循环,包括在98°C变性15 s,在55°C退火5 s和在72°C引物延伸20 s。然后使用Plus DNA Clean / Extraction Kit(GMbiolab Co,Ltd.#DP034P)通过凝胶切割纯化PCR产物,并在30μLddH 2 O中洗脱。
对于509寡核苷酸池装配片段的制备,我们从如上所述的主池开始。使用Q5®高保真DNA聚合酶和相应的引物(不同序列在补充表1中显示),用不同的同源臂制备片段 。模板使用来自主库的0.2 ng寡核苷酸输入进行50μLPCR反应。初始变性在98°C进行5分钟。然后进行20个PCR循环,包括在98°C变性30 s,在56°C退火30 s和在72°C引物延伸20 s。最后,将溶液在72°C下孵育5分钟以终止PCR反应。然后使用Plus DNA Clean / Extraction Kit(Gmbiolab,#DP034P)纯化文库,并在30μLddH 2中洗脱O.最终库在与上述相同的条件下运行。然后根据用户手册使用GibsonAssembly®预混料-组合件(NEB,#E2611)。
对于11520寡核苷酸池装配片段的制备,我们从如上所述的主池开始。使用2xEasyTaq®PCR SuperMix(AS111,TRANS)和相应的引物,在不同的同源臂上制备片段(序列显示在补充表 1和补充图 4中)。该模板将来自主库的20 ng寡核苷酸输入用于50μLPCR反应中。初始变性在94°C下进行2分钟。随后进行10个PCR循环,包括在94°C变性30 s,在53°C退火30 s和在72°C引物延伸20 s。最后,将溶液在72°C下孵育5分钟以终止PCR反应。然后纯化文库并在100μLddH 2中洗脱O.根据用户手册使用NEBuilder®HiFi DNA组装克隆试剂盒(NEB,#E5520)。
以上仅为部分内容摘录,该文自然杂志地址:https://www.nature.com/articles/s42003-020-01141-7
声明: 此文观点不代表本站立场;转载须要保留原文链接;版权疑问请联系我们。