一种结合随机访问、DNA 组装和纳米孔测序的 DNA 存储方法成功恢复了存储在 111,499 个寡核苷酸中的数字信息,总计 1.67 TB 的数据。与之前在 DNA 存储系统中使用纳米孔的报告相比,该方法可使测序和解码能力提高约 100 倍。
华北电力大学 控制与计算机工程学院,的宋子臣等三名学生在MDPI期刊上发布了一篇关于DNA存储相关的文章,翻译如下:
摘要
脱氧核糖核苷酸(DNA)分子是大量遗传信息的稳定载体,为下一代信息处理技术提供了理想的存储介质。处理 DNA 信息的技术代表了生物学和计算机技术的跨学科整合,已经成为单独处理电子信息的技术的有吸引力的替代品。DNA技术的具体应用可以分为三个部分:存储、计算和自组装。DNA 信息处理的质量取决于 DNA 读取的准确性。纳米孔检测使研究人员能够准确地对核苷酸进行测序,因此被广泛用于读取 DNA。在本文中,我们介绍了纳米孔检测的原理和发展历史,并对涉及纳米孔检测和基于纳米孔的存储的 DNA 信息处理的最新进展和具体应用进行了系统回顾。我们还讨论了人工智能在纳米孔检测和 DNA 信息处理方面的潜力。该工作不仅为未来纳米孔检测的发展提供了新的途径,也为构建更先进的DNA信息处理技术奠定了基础。
关键词:
纳米孔检测;DNA储存;ONT纳米孔;人工智能;DNA信息处理
一、简介
电子信息存储系统是现代信息技术的基本组成部分。然而,随着大数据时代的到来,存储容量往往无法满足系统需求。电子信息的激增 [ 1 ] 导致了基于磁体和半导体的大数据长期存储系统的发展 [ 2 ]。这些系统具有不可持续的缺点,包括有限的寿命 [ 3 ]、高昂的基础设施成本和巨大的功耗 [ 4 ]。DNA作为遗传信息的生物存储载体,是下一代存储系统的理想介质,具有以下优势:超高信息密度(455EB数据/克[5 ]); 寿命长(半衰期 > 500 年 [ 6 , 7 ]);相对较低的能源消耗;可编程性;和可寻址性 [ 8 ]。因此,处理 DNA 信息的技术受益于生物学和信息技术之间的跨学科整合 [ 9 ]。这些技术的三个基本组成部分——DNA 计算、DNA 存储和 DNA 自组装——为处理大量信息提供了一种新方法。
分子信息的解码对于 DNA 信息处理至关重要,DNA 存储就是一个典型的例子。在 DNA 存储中,数字信息可以按照特定的算法编码成 DNA 序列,然后通过核苷酸合成技术存储在 DNA 链中,并通过 DNA 测序的方法读出。DNA 存储的质量取决于 DNA 合成和测序[ 10、11 ]。大规模的 DNA 可以通过快速低成本的固相合成法合成 [ 12 , 13 ]。传统的 DNA 测序方法,例如 Sanger 测序 [ 14 ]] 和 Illumina 测序,成本高昂,可能导致大规模 DNA 存储失败。因此,需要一种高效的DNA测序方法来支持DNA信息处理商业应用的扩展。
纳米孔检测是一种单分子检测技术,可用于单分子化学研究 [ 15 ]、肽和蛋白质折叠研究 [ 16、17 ] 、与核酸或核酸-蛋白质解链相关的机械方面的分析配合物 [ 18 , 19 , 20 ]。在DNA信息处理领域,纳米孔检测是一种高效的检测技术,主要有两个应用:DNA纳米孔测序和单分子传感[ 21 ]。利用单核苷酸分辨率的纳米孔测序技术从DNA链中读取信息,从而辅助大规模DNA存储系统的无错数据恢复[22 , 23 ]。此外,可以组装纳米孔作为用于分子识别的单分子传感器。通过修饰生物纳米孔,可以提高纳米孔的特异性和灵敏度,从而扩大分子的检测范围。目前,纳米孔可以检测多种分子,包括 DNA [ 24、25、26、27 ] 、具有损伤和核苷酸修饰的DNA和RNA [ 28、29、30、31、32、33 ]、全细胞核酸酸制剂 [ 34 , 35 , 36, 37 , 38 ], 未折叠蛋白肽 [ 39 , 40 , 41 , 42 , 43 , 44 , 45 , 46 ] 和细菌毒素 [ 47]. 由于纳米孔具有高效的分子识别能力,DNA数据库在信息存储方面有了更多的选择。以DNA修饰为例。DNA 修饰经过生物化学处理以将 DNA 链与其他分子结合。对于相同的 DNA 修饰,修饰核苷酸的存在或不存在可以被认为是沿着寡核苷酸链的数字位“1”和“0”。因此,DNA修饰序列在逻辑上可以看作是二元序列。另一方面,纳米孔可以精确识别和区分多种DNA修饰,扩展了DNA存储的字母表,直接提高了DNA存储的潜在存储密度。目前,纳米孔检测技术已广泛应用于DNA存储系统。
本综述旨在涵盖纳米孔检测技术的最新进展及其在 DNA 信息处理中的应用。为此,我们对以下内容进行了系统的研究:(1)纳米孔检测技术的原理和发展历程;(2) 纳米孔检测的进展;(3) 两种利用纳米孔检测的DNA存储方式;(4)人工智能(AI)在纳米孔数据处理和DNA信息技术中的应用。我们设想这篇评论文章将推动 DNA 信息处理领域纳米孔检测的发展。
2. 单分子检测的原理和历史
纳米孔检测是一种单分子检测技术,起源于1976年的膜片钳技术[ 48 ]。单分子检测具有实时监测的特点,可用于获取分子的功能、结构、动力学等信息[ 49 ]。根据检测原理,单分子技术的方法学分为光学、机械和电化学三大类。光学方法给分子添加荧光生物标记,通过显微镜观察荧光的激发或猝灭[ 50 ]。然而,这些方法会改变分子结构,从而导致观察结果不准确。机械方法使用原子力显微镜 [ 51] 或扫描隧道显微镜 [ 52 ] 来拍摄生物分子结构,并且可以提供没有荧光标记的生物分子的直接图像。不幸的是,机械方法有几个缺点使其不适用于大规模分子检测:即笨重的观察设备、高成本和复杂的操作。基于电化学的单分子检测技术通常依靠灵敏的电流监测器通过分析离子电流信号来检测分子[ 53 ];纳米孔检测技术就是其代表之一。
2.1. 生物纳米孔检测技术的一般原理
近年来,纳米孔检测以其无需荧光标记、设备便携(Minion仅90 g[ 54 ])、检测成本低[ 55 ]等优势受到了研究人员的广泛关注。纳米孔检测实验仪器的工作原理类似于库尔特计数器 [ 50 ],单个带电分子在电场力的作用下穿过嵌入膜中的微小孔,产生瞬态电流阻断信号。生物纳米孔检测的一般流程 [ 56 , 57] 首先将生物纳米孔嵌入电阻聚合物膜中。装有电解质溶液的罐被膜分隔成两个腔室,纳米孔成为两个腔室之间的唯一通道。在腔室中添加两个电极,并向电极施加恒定电压;负电压侧定义为“cis”,相反侧定义为“trans”。在检测过程中,感兴趣的分子在施加的电场或/和随后出现的离子选择性纳米孔内的电渗流的影响下沿着纳米孔被驱动[ 58]. 带电的生物分子可以从“顺式”到“反式”或相反方向驱动通过纳米孔。由于纳米孔的纳米级内径,通过纳米孔的生物分子会阻碍离子电流的流动,产生离散的信号变化。通过使用计算机算法分析离子电流的信号特征,可以得出有关目标生物分子的各种直接信息,例如其种类、结构和功能。
2.2. 纳米孔检测技术的发展
在纳米孔检测的发展过程中,DNA或RNA测序最初的重点是实现高质量的碱基检出。DNA 纳米孔测序的概念具体起源于 80 年代后期。1989年,Hagan Bayley的团队开始探索α-溶血素等寡聚跨膜蛋白孔的结构和功能[ 59 ],随后他们假设通道蛋白可以作为分子的生物传感器[ 60 ]。1996 年,Kasianowicz 等人。是第一组通过 α-溶血素纳米孔捕获单链 DNA (ssDNA) 易位产生的离子电流信号 [ 61],并且在 1999 年,Akeson 等人。利用 α-溶血素作为生物传感器,可以快速区分 RNA 分子中的嘧啶和嘌呤片段 [ 62 ]。十多年后,Gundlach 等人。使用耻垢分枝杆菌孔蛋白 A (MspA) [ 63 ] 作为生物纳米孔,结合噬菌体 phi29 DNA 聚合酶,将可识别的 DNA 碱基数量增加到约 30 [ 64 ]。随后,2014年,Oxford Nanopore Technologies(ONT)推出了第一款商用纳米孔测序仪MinION,具有单分子检测、读长长、测序速度快、便携等优点[ 54 ]。
2.3. 纳米孔的分类
目前,根据原料组成,纳米孔有两类:蛋白质纳米孔和固体纳米孔(由固体材料制成)。由于其相对较小的内径,生物纳米孔具有高信噪比和分辨率。此外,随着蛋白质定点修饰的出现,生物纳米孔可用于检测范围广泛的分子,包括 DNA [ 65 ]、RNA [ 66 ]、蛋白质和金属离子 [ 67 ]。生物纳米孔包括 α-溶血素 (α-HL)、MspA、大肠杆菌溶细胞素 (ClyA) [ 68 ] 和气溶素 [ 69 ]。然而,蛋白质纳米孔的结构稳定性容易受到环境条件的影响。
与生物纳米孔相比,固态纳米孔具有优异的几何灵活性、机械和化学稳定性以及与现代半导体和微流体制造技术兼容的特性[ 70、71、72、73、74、75 ]。用于制备固态纳米孔的材料包括无机硅[ 76 ]、玻璃毛细管[ 77 ]和石墨烯[ 78 ]]. 其中,氮化硅是应用最广泛的材料,因为它具有较低的机械应力和优异的化学稳定性。尽管固态纳米孔的优异性能在单分子水平的生物分子检测中具有巨大潜力,但它们仍然受到许多特殊缺点的困扰,包括成本高[ 79 ],不同纳米孔之间的重现性差,内径大,缺乏原子分辨率功能化 [ 80 ]。
3. 纳米孔检测 DNA 分子携带的特定修饰
纳米孔可用于同时检测有关单链/双链 DNA 在蛋白质结合或无机化学修饰过程中发生的多种变化或损伤的信息[ 81、82 ]。
3.1. 检测 DNA 损伤
未修复的 DNA 损伤的积累可能导致细胞过早衰老、癌症和一些神经退行性疾病[ 83、84 ]。目前,纳米孔被用作 DNA 损伤检测的生物传感器。2019 年,Ma 等人。提出了一种纳米孔检测方法来识别 DNA 上的顺铂损伤 [ 85 ](图 1a)。顺铂与N 7结合嘌呤原子在 DNA 上形成损伤,抑制 DNA 在癌细胞中的正常复制和转录。马等。应用 MspA 准确检测顺铂诱导的 DNA 损伤,证明纳米孔测序技术可以在小于 10 ng 的输入测序文库中识别顺铂损伤。此外,通过观察 DNA 通过纳米孔的易位速度,可以区分多个 DNA 损伤。2022 年,张等人。通过观察酶促减速的动力学,实现了对 O6-羧甲基鸟嘌呤 (O6-CMG)、O6-甲基鸟嘌呤 (O6-MG) 和无碱 (AP) 位点的直接鉴定 [ 86 ](图 1b). 他们观察到 phi29 DNA 聚合酶的渐进运动受到 DNA 损伤(如 O6-CMG)的阻碍,并记录了酶促停滞,表明运动酶和 DNA 损伤之间相互作用产生的动力学信息可用于识别多个 DNA 损伤。
3.2. 核酸检测
当前的纳米孔测序技术能够检测异源核酸。异源核酸,也称为异种核酸 (XNA),是一类具有非天然主链或核碱基的核酸分子 [ 87 , 88 , 89 ]。2019 年,Yan 等人。提出了一种通过纳米孔诱导相移测序 (NIPSS) 对 2-脱氧-2-氟阿拉伯酸 (FANA) 进行直接测序的方法 [ 90 ](图 1C)。通过将 FANA 与 DNA 驱动链连接,他们表明可以通过使用 phi29 DNA 聚合酶的 NIPSS 实现 FANA 的直接测序。他们的贡献促成了一种基于纳米孔测序的通用鉴定方法的发展,该方法可用于清楚地区分 DNA、RNA 和 XNA 核苷酸。
3.3. 多肽和蛋白质的检测
也可以使用纳米孔测序技术实现蛋白质检测 [ 20 , 91 , 92 ]。2021 年,Wang 等人。观察到肽与 MspA 的单分子棘轮效应 [ 93 ](图1d)。通过构建肽-寡核苷酸偶联物 (POC) 并使用 NIPSS 进行测量,他们直接观察了肽棘轮运动的离散步骤。实验上,NIPSS 测量产生的肽的电流信号结果显示出高度一致的模式,与肽的氨基酸序列具有明显的相关性。随后,在 2021 年 11 月,Brinkerhoff 等人。展示了一种基于纳米孔的肽测序方法,具有单氨基酸分辨率 [ 94 ]]. 他们设计了一个实验,在这个实验中,POC 被 DNA 解旋酶驱动通过 MspA 纳米孔,并且他们能够观察到由肽棘轮通过纳米孔产生的阶梯状电流信号。此外,他们改变了肽序列上固定位点的氨基酸,并观察了由此产生的离子电流序列变化,从而实现了蛋白质测序。
3.4. 检测无机化学分子
除了检测生物分子外,纳米孔还可用于检测无机化学分子。2021 年,Jia 等人。提出了一种基于纳米孔测序技术的用于随机随机传感 (PNRSS) 的可编程纳米反应器,能够在单分子水平上实时监测化学反应 [ 95 ](图1e)。通过 PNRSS 直接观察到金属离子、简单有机化合物(如乳酸)和核苷类似物的各种单分子反应。此外,他们使用人工智能工具来提高 PNRSS 的传感分辨率,最终使他们能够检测到总共 20 种化学反应。
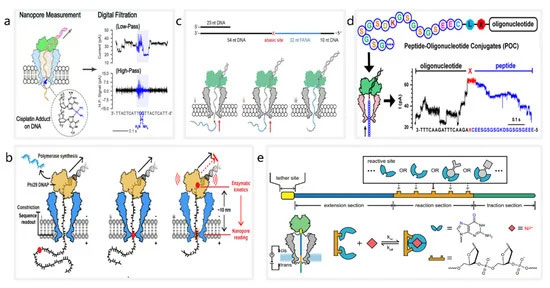
图 1. 基于纳米孔的分子检测系统示意图。( a ) 检测 DNA 上的顺铂损伤。经 ACS Sensors Fubo Ma 许可转载;由美国化学学会出版,2021 年。( b ) 使用 MspA 纳米孔(蓝色)和 phi29 DNAP(黄色)在纳米孔测序过程中的停滞动力学读数。经 Jinyue Zhang 许可转载,Nano Letters;由美国化学学会出版,2022 年。( c ) 嵌合 DNA(灰色)-FANA(青色)与无碱基间隔区(红色)的测序。转载自 [ 90 ]。( d ) 使用 NIPSS 的 POC 棘轮运动。转载自颜双红,Nano Letters;美国化学学会,2021 年。(e) 通过PNRSS检测无机化学分子。转载自 [ 95 ]。
4. 基于纳米孔测序技术的 DNA 存储
虽然携带数字信息的核苷酸链很脆弱,在保存过程中可能会受到外界环境因素的干扰和破坏,如紫外线、极端温度变化、细菌、病毒等生物污染等。然而,DNA具有超高信息密度和长寿命等特性,有望成为下一代信息处理系统的新型数据存储介质。我们有理由相信,随着DNA保存技术和保存设备的进步[ 96 , 97 , 98 , 99】,DNA存储的容量、持续时间和存储质量将得到极大提升。目前,DNA纳米孔可用于鉴定DNA/RNA的序列和DNA/RNA的分子或化学修饰,这些都可以被视为位点,从而为DNA存储系统提供更多选择。作为数字数据存储载体的DNA有两类:(1)人工合成的DNA碱基序列;(2) DNA 纳米结构/修饰。
4.1. 基于合成 DNA 序列的 DNA 存储
如图2a所示,DNA存储的整个过程通常分为六个步骤[ 100 ](图2a):(1)将数字信息编码成DNA序列;(2)设计和合成DNA序列;(3) 体内或体外保存DNA;(4) 特定DNA序列的随机存取;(5) 特定DNA序列的读取;(6) 将 DNA 序列解码并恢复为数字信息。目前,纳米孔测序技术广泛应用于步骤4和5。
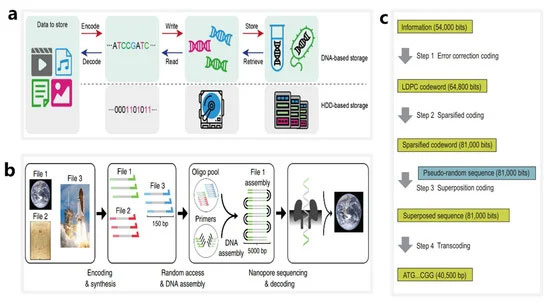
图 2. 基于合成核苷酸序列的 DNA 存储系统的工作流程。( a ) 与传统硬盘存储相对应的 DNA 中数字数据存储的主要步骤。转载自 Yaya Hao,SMALL STRUCTURES;由 John Wiley and Sons 出版,2020 年。( b ) 使用 ONT 纳米孔作为工具对通过随机访问获得的长双链 DNA 链进行测序的 DNA 数据存储工作流程概述。转载自 [ 101 ]。( c ) 使用人工酵母染色体的体内 DNA 存储系统的工作流程。转载自 [ 22 ]。
纳米孔测序技术有利于DNA存储系统的数据解码。2018 年,Lee 等人。使用存储在 35 个文件中的超过 1300 万个寡核苷酸和总计 200 MB 的数据设计并验证了一个大型引物库,他们能够使用随机访问方法和纳米孔测序实现所有 DNA 文件的无错误数据恢复 [ 23 ]。2019 年,洛佩兹等人。展示了一种结合随机访问、DNA 组装和纳米孔测序的 DNA 存储方法 [ 101 ](图 2b). 他们使用 MinION 测序仪成功恢复了存储在 111,499 个寡核苷酸中的数字信息,总计 1.67 TB 的数据。与之前在 DNA 存储系统中使用纳米孔的报告相比,该方法可使测序和解码能力提高约 100 倍。
纳米孔测序技术可应用于体内和体外存储系统。2021 年,Chen 等人。使用 254,886 bp 的人工酵母染色体设计并合成了体内系统 [ 22 ](图2c)。使用稀疏低密度奇偶校验 (LDPC) 码和伪随机序列将染色体写入 37,782 位数据,总共包括两张图像和一段视频剪辑。在DNA信息读取阶段,他们使用纳米孔测序仪实现精准碱基调用,以10.79%的错误率实现了可靠的数据恢复。
4.2. 使用DNA纳米结构和修饰作为信息载体的DNA存储
纳米孔测序技术可准确识别 DNA 修饰或纳米结构,为 DNA 存储提供了新的解决方案。高度可编程的 DNA 纳米结构提供了多种用于存储数字数据的地址位点[ 102、103 ]。2018 年,Chen 和 Kong 等人。提出了一种将 DNA 发夹视为位点的 DNA 存储方案 [ 104 ](图 3一种)。在他们的工作中,不同长度的 DNA 发夹被视为数字位,用于开发高分辨率固态纳米孔测序方法。使用内径约为 5 nm 的石英纳米孔可以清楚地区分长度为 8 bp 和 16 bp 的 DNA 发夹。因此,8-bp和16-bp的发夹分别被指定为bit-0和bit-1,用于将56个发夹连接到7228-bp长的寡核苷酸上,从而形成56-bit的存储片段。使用类似的想法,Bell 和 Keyser [ 105]根据DNA折纸原理设计了一个DNA纳米结构库,其中每个成员都有一个唯一的条形码,条形码上的每一位都由一个DNA哑铃发夹的存在与否来表示。他们最终证实,可以通过固态纳米孔测序以 94% 的准确率识别 3 位条形码。
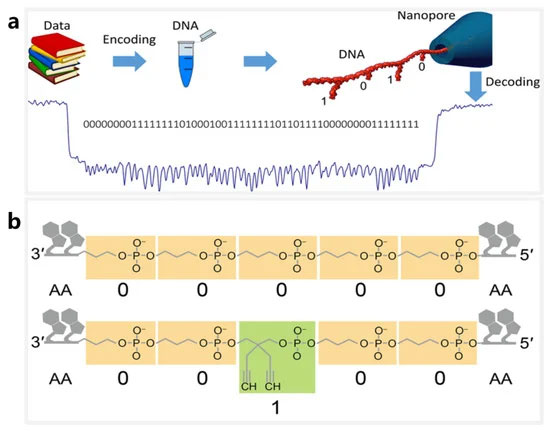
图 3. 基于 DNA 纳米结构的分子存储系统。( a ) 使用纳米孔测量 DNA 载体的示意图,其中位“1”和“0”分别代表 16 bp 和 8 bp 的 DNA 发夹结构。经 Kaikai Chen 许可转载,Nano Letters;美国化学学会,2019 年。( b ) 生物聚合物序列图解,其中“0”代表单体分子,“1”代表其甲基化形式。转载自 [ 106 ]。
纳米孔的替代 DNA 存储系统正在用于识别生物聚合物序列。2020 年,Cao 等人。使用专门定制的生物聚合物序列作为位信息存储载体[ 106 ](图3b). 生物聚合物序列是生物杂化大分子,包含两种不同大小的单体(正丙基磷酸酯和 [2,2-二炔基]-丙基磷酸酯)和天然核苷酸,其中单体分别映射为 bit-0 和 bit-1。该研究使用气溶素毒素的生物工程纳米孔,以单碱基分辨率成功实现了定制生物聚合物的位点识别。此外,还应用深度学习实现了高达4位数字序列的高精度编解码。这个独特的系统为开发新的 DNA 存储系统提供了灵感。
5. 人工智能在纳米孔数据处理和DNA信息技术中的应用
近年来,人工智能与DNA信息处理的结合取得了意想不到的好结果。深度学习是人工智能的一个分支,主要使用多层神经网络从数据中学习。深度学习模型可以对原始数据进行自动学习和特征提取,具有强大的挖掘大数据潜在规律的能力。诸如卷积神经网络 (CNN)、深度置信网络和递归神经网络 (RNN) 等深度学习模型已经开发出来,它们在多项任务上表现良好,包括计算机视觉、语音识别和自然语言处理[ 107、108、109 ]。根据之前的报道 [ 110 , 111],纳米孔检测产生的海量序列数据导致了多种深度学习模型的设计和训练,这些模型正在广泛应用于DNA信息处理任务,如碱基调用、生物分子检测和DNA存储。
5.1. 碱基检出
碱基调用是在测序过程中推断 DNA 片段中核苷酸顺序的过程 [ 112 ]。由于纳米孔测序会产生电流信号,因此碱基检出需要计算机算法来处理序列数据。迄今为止,包括 ONT 团队在内的许多研究人员已经设计了多种基于深度学习模型的软件程序来进行碱基识别。这些软件程序可以按两种类型的输入数据分类:分段事件和原始电流信号。
早期的碱基识别软件依赖于对分段事件的分析。2016 年,David 等人的研发团队开发了 Nanocall,这是第一个用于牛津纳米孔测序数据的开源离线碱基识别器 [ 113 ]。它使用基于隐马尔可夫模型 (HHM) 进行碱基序列识别。使用 R7.3 版本的 MinION,Nanocall 分析了来自两个大肠杆菌和两个人类基因样本的数据,发现具有 68% 同一性的读数。由于 HHM 不适用于长均质聚合物检测,因此将 RNN 应用于分段事件序列。2016 年,Boza 等人。提出了 DeepNano,一种具有深度 RNN 的开源 DNA 碱基调用程序 [ 114 ]。使用大肠杆菌和肺炎克雷伯菌的 R7.3 测试数据集为了评估 DeepNano 的碱基识别精度,他们发现,对于 2D 读取,DeepNano 的碱基识别精度分别达到 88.5% 和 86.7%。在base calling的速度上,DeepNano比Nanocall快5到20倍。
通过深度学习模型将原始电流信号直接转换为碱基序列既方便又准确。BasecRAWller 是一种基于原始电流信号的纳米孔数据碱基调用软件,由 Stoiber 等人提出。2017 年 [ 115 ]。BasecRAWller 使用一对单向 RNN 直接从原始纳米孔读数中进行实时 DNA 碱基调用,并根据其性能使用两个数据集进行了评估:大肠杆菌和人类。据报道,BasecRAWller 读取在大肠杆菌和人类数据集上具有 81.7% 和 72.5% 的同一性。此后不久,滕等人。报道了 Chiron,这是第一个实现端到端碱基调用并将原始电流信号直接转换为核苷酸序列的深度学习模型 [ 116]. Chiron 将 CNN 与 RNN 和联结时间分类解码器相结合,使其能够直接从原始信号数据中学习,而无需使用事件分割。Chiron 在大肠杆菌和人类数据集上的识别率分别为90.57% 和 81.54% ,高于上述其他三个软件程序。就中央处理器的速度而言,Chiron 比 BasecRAWller 慢(分别为 21 bp/s 和 81 bp/sec)。此外,Chiron 是完全开源的,允许用户训练自己的神经网络并开发专门的碱基识别应用程序。
5.2. 生物分子检测
深度学习是提高多重生物分子纳米孔识别精度的有力工具,可有效应用于生物分子修饰检测。2019 年,刘等人。设计并训练了 DeepMod [ 117 ](图4a ),这是一种具有短期长期记忆 (LSTM) 的双向 RNN 模型,适用于从 ONT 纳米孔测序仪提取的原始电流信号中进行高精度 DNA 修饰检测。DeepMod 在大肠杆菌、衣藻的纳米孔读数中进行了评估reinhardtii 和人类基因组。结果表明,DeepMod可以高精度检测甲基化修饰的DNA核苷酸;例如,5-甲基胞嘧啶 (5 mC) 和 6-甲基腺嘌呤 (6 mA) 的平均检测准确度分别为 0.99 和 0.9。同样,在 2021 年,Ni 等人。设计了一种称为 DeepSignal-plant 的深度学习工具,该工具使用纳米孔读数对植物中自然发生的胞嘧啶甲基化的所有三个序列背景进行全基因组检测 [ 118 ](图4b)。DeepSignal-plant 采用多层双向 RNN 架构,后跟全连接层,可以自动从纳米孔数据中提取和学习序列特征和信号特征,其大小是其前身 DeepSignal 的八分之一 [119 ]。
图 4. AI 在 DNA 信息存储中的应用。( a ) 神经网络模型 DeepMod 的架构,用于捕获纳米孔信号的时间序列特征并检测 DNA 修饰。转载自 [ 117 ]。( b ) DeepSignal-plant 的架构,其中可以使用双向 LSTM (biLSTM) 提取序列和信号特征。转载自 [ 118 ]。( c ) 混合预测 DNA 编码的进化模型工作流程。转载自 [ 120 ]。( d ) 用于快速读取分子标签的 CNN 示意图。转载自 [ 121 ]。
5.3. DNA储存
人工智能可以集成到DNA存储的多个环节,有可能加快寡核苷酸的读取速度,提供高效准确的随机存取方法,推动大规模DNA存储系统商业化的进一步实现。
深度学习在快速分析纳米孔读数方面具有巨大潜力。2020 年,Nivala 等人。提出了一种在射频识别标签和快速响应代码等传统方法不适用的情况下使用 DNA 或其他分子标记物理对象的方法 [ 120 ](图4c)。他们开发了 Porcupine 系统,这是一种最终用户分子标记系统,能够使用便携式纳米孔设备在几秒钟内读取基于 DNA 的标记。它的数字位由不同 DNA 链的存在或不存在表示,称为分子位 (molbits),由 CNN 直接从原始纳米孔信号中分类。这种方法避免了使用 DNA 序列进行碱基调用的需要,从而大大减少了时间要求和复杂性。
综上所述,深度学习可以应用于快速提高DNA测序的碱基识别准确率(同一性从68%提高到90.57%),极大地扩展了纳米孔可检测的分子类型范围,为高分子测序提供了可能。加快 DNA 读取速度,所有这些都是 DNA 存储和纳米孔技术大规模商业化的关键因素。
深度学习还可以用于通过计算机模拟扩展 DNA 存储系统的容量,这为指导研究提供了有益的建议。能够使用大规模的 DNA 存储系统相当于能够设计复杂的 DNA 引物序列,这在大多数研究环境中是负担不起的。因此,DNA杂交过程的精确控制和预测对于大规模DNA存储系统的设计至关重要。2021 年,David Buterez 率先提出了用于 DNA 杂交预测的机器学习技术的综合研究 [ 121 ](图4d)。作为基准,他在计算机上对多个机器学习模型进行了性能评估-生成的杂交数据集包含超过 250 万个 DNA 序列对。接下来,他使用 CNN、RNN 和 RoBERTa 模型评估了这个数据集,发现深度学习模型提供了更准确的 DNA 杂交预测,并且与基线模型相比,运行时间减少了一到两个数量级。
6.结论
DNA信息处理技术利用DNA分子作为数据存储介质和数据计算单元,具有存储大数据的潜力。然而,这些技术的发展一直受到传统 DNA 测序成本高、速度慢、准确性相对较低的阻碍。纳米孔检测是一种新的单分子检测技术,通过分析分子通过纳米孔时产生的离子电流信号,实现分子鉴定。与其他方法相比,快速纳米孔检测具有免标记、低成本、方便等优点,满足了DNA信息处理的要求。目前,纳米孔检测广泛应用于DNA信息处理任务,如生物分子检测、DNA存储等。然而,
由于特异性和灵敏度的提高,纳米孔经常用于生物分子检测,并且可用于检测各种分子,包括核酸、蛋白质和无机离子。然而,性能优异的纳米孔检测平台尚未系统统一,以消除不同实验室的重复实验。事实上,多个研究团队已经独立设计了不同的生物纳米孔来检测分子。由于缺乏一致的标准,同一分子的检测结果报告范围很广,可能会阻碍生物分子检测的发展。
在 DNA 存储系统中,数字信息可以存储在 DNA 序列、包含修饰 DNA 的序列或其他生物分子中。纳米孔技术可以优化DNA信息的读取过程,为DNA存储领域提供高速、准确的碱基调用。然而,DNA 纳米孔测序在两个方面仍然受到限制。首先,虽然纳米孔测序的碱基识别准确率有了很大提高,目前在 90-95% 之间,但仍低于下一代测序的 99% 准确率 [ 122 ]]. 其次,纳米孔检测的通量相对较低。纳米孔检测依赖于分子通过纳米孔的微电流,同时检测多个纳米孔会因电信号叠加而导致信号失真。目前单个纳米孔的读取速度约为 10 毫秒/碱基,这将需要大约 20 年的时间才能以 10 倍覆盖深度对人类基因组进行测序 [ 123 ]。因此,更高的测序精度和检测通量可以促进大规模DNA存储的更广泛商业应用。
人工智能在纳米孔技术中的应用有望克服上述障碍。在生物分子检测中,深度学习模型中模式识别的强大功能可以同时检测多种分子。此外,AI 提供高精度序列预测,从而提高 DNA 存储的碱基调用速度和准确性。最后,人工智能还可以有效地预测蛋白质的分子折叠,这有助于修饰生物纳米孔的结构,可能为提高检测通量提供新途径。借助更先进的纳米技术和人工智能,我们预计DNA纳米孔将继续在DNA信息处理领域提供新的应用。
References
- Zhirnov, V.; Zadegan, R.M.; Sandhu, G.S.; Church, G.M.; Hughes, W.L. Nucleic acid memory. Nat. Mater. 2016, 15, 366–370. [Google Scholar] [PubMed]
- Goda, K.; Kitsuregawa, M. The History of Storage Systems. Proc. IEEE Inst. Electr. Electron. Eng. 2012, 100, 1433–1440. [Google Scholar]
- An?el, A.; Heider, D.; Hattab, G. The visual story of data storage: From storage properties to user interfaces. Comput. Struct. Biotechnol. J. 2021, 19, 4904–4918. [Google Scholar] [PubMed]
- Andrae, A.S.G.; Edler, T. On Global Electricity Usage of Communication Technology: Trends to 2030. Challenges 2015, 6, 117–157. [Google Scholar]
- Church, G.M.; Gao, Y.; Kosuri, S. Next-generation digital information storage in DNA. Science 2012, 337, 1628. [Google Scholar] [CrossRef]
- Allentoft, M.E.; Collins, M.; Harker, D.; Haile, J.; Oskam, C.L.; Hale, M.L.; Campos, P.F.; Samaniego, J.A.; Gilbert, M.T.; Willerslev, E.; et al. The half-life of DNA in bone: Measuring decay kinetics in 158 dated fossils. Proc. Biol. Sci. 2012, 279, 4724–4733. [Google Scholar]
- Bhat, W.A. Bridging data-capacity gap in big data storage. Future Gener. Comput. Syst. 2018, 87, 538–548. [Google Scholar]
- Jaeger, L.; Chworos, A. The architectonics of programmable RNA and DNA nanostructures. Curr. Opin. Struct. Biol. 2006, 16, 531–543. [Google Scholar]
- Ishmukhametov, I.; Batasheva, S.; Rozhina, E.; Akhatova, F.; Mingaleeva, R.; Rozhin, A.; Fakhrullin, R. DNA/Magnetic Nanoparticles Composite to Attenuate Glass Surface Nanotopography for Enhanced Mesenchymal Stem Cell Differentiation. Polymers 2022, 14, 344. [Google Scholar] [CrossRef]
- Dong, Y.; Sun, F.; Ping, Z.; Ouyang, Q.; Qian, L. DNA storage: Research landscape and future prospects. Natl. Sci. Rev. 2020, 7, 1092–1107. [Google Scholar]
- Heckel, R.; Shomorony, I.; Ramchandran, K.; David, N. Fundamental limits of DNA storage systems. In Proceedings of the 2017 IEEE International Symposium on Information Theory (ISIT), Aachen, Germany, 25–30 June 2017; pp. 3130–3134. [Google Scholar]
- Merrifield, B. Solid phase synthesis. Science 1986, 232, 341–347. [Google Scholar] [PubMed]
- Belitsky, J.M.; Nguyen, D.H.; Wurtz, N.R.; Dervan, P.B. Solid-phase synthesis of DNA binding polyamides on oxime resin. Bioorg. Med. Chem. 2002, 10, 2767–2774. [Google Scholar] [PubMed]
- Sikkema-Raddatz, B.; Johansson, L.F.; de Boer, E.N.; Almomani, R.; Boven, L.G.; van den Berg, M.P.; van Spaendonck-Zwarts, K.Y.; van Tintelen, J.P.; Sijmons, R.H.; Jongbloed, J.D. Targeted next-generation sequencing can replace Sanger sequencing in clinical diagnostics. Hum. Mutat. 2013, 34, 1035–1042. [Google Scholar] [PubMed]
- Cao, C.; Long, Y.-T. Biological Nanopores: Confined Spaces for Electrochemical Single-Molecule Analysis. Acc. Chem. Res. 2018, 51, 331–341. [Google Scholar]
- Oukhaled, A.; Bacri, L.; Pastoriza-Gallego, M.; Betton, J.-M.; Pelta, J. Sensing Proteins through Nanopores: Fundamental to Applications. ACS Chem. Biol. 2012, 7, 1935–1949. [Google Scholar]
- Mereuta, L.; Asandei, A.; Seo, C.H.; Park, Y.; Luchian, T. Quantitative Understanding of pH- and Salt-Mediated Conformational Folding of Histidine-Containing, β-Hairpin-like Peptides, through Single-Molecule Probing with Protein Nanopores. ACS Appl. Mater. Interfaces 2014, 6, 13242–13256. [Google Scholar]
- Hornblower, B.; Coombs, A.; Whitaker, R.D.; Kolomeisky, A.; Picone, S.J.; Meller, A.; Akeson, M. Single-molecule analysis of DNA-protein complexes using nanopores. Nat. Methods 2007, 4, 315–317. [Google Scholar]
- Mathé, J.; Visram, H.; Viasnoff, V.; Rabin, Y.; Meller, A. Nanopore unzipping of individual DNA hairpin molecules. Biophys. J. 2004, 87, 3205–3212. [Google Scholar] [CrossRef]
- Luchian, T.; Park, Y.; Asandei, A.; Schiopu, I.; Mereuta, L.; Apetrei, A. Nanoscale Probing of Informational Polymers with Nanopores. Applications to Amyloidogenic Fragments, Peptides, and DNA–PNA Hybrids. Acc. Chem. Res. 2019, 52, 267–276. [Google Scholar] [CrossRef]
- Desai, T.A.; Hansford, D.J.; Kulinsky, L.; Nashat, A.H.; Rasi, G.; Tu, J.; Wang, Y.; Zhang, M.; Ferrari, M. Nanopore technology for biomedical applications. Biomed. Microdevices 1999, 2, 11–40. [Google Scholar] [CrossRef]
- Chen, W.; Han, M.; Zhou, J.; Ge, Q.; Wang, P.; Zhang, X.; Zhu, S.; Song, L.; Yuan, Y. An artificial chromosome for data storage. Natl. Sci. Rev. 2021, 8, nwab028. [Google Scholar] [CrossRef]
- Organick, L.; Ang, S.D.; Chen, Y.-J.; Lopez, R.; Yekhanin, S.; Makarychev, K.; Racz, M.Z.; Kamath, G.; Gopalan, P.; Nguyen, B.; et al. Random access in large-scale DNA data storage. Nat. Biotechnol. 2018, 36, 242–248. [Google Scholar] [CrossRef]
- Schneider, G.F.; Dekker, C. DNA sequencing with nanopores. Nat. Biotechnol. 2012, 30, 326–328. [Google Scholar] [CrossRef] [PubMed]
- Goto, Y.; Akahori, R.; Yanagi, I.; Takeda, K.-i. Solid-state nanopores towards single-molecule DNA sequencing. J. Hum. Genet. 2020, 65, 69–77. [Google Scholar] [CrossRef] [PubMed]
- Clarke, J.; Wu, H.-C.; Jayasinghe, L.; Patel, A.; Reid, S.; Bayley, H. Continuous base identification for single-molecule nanopore DNA sequencing. Nat. Nanotechnol. 2009, 4, 265–270. [Google Scholar] [CrossRef] [PubMed]
- Traversi, F.; Raillon, C.; Benameur, S.M.; Liu, K.; Khlybov, S.; Tosun, M.; Krasnozhon, D.; Kis, A.; Radenovic, A. Detecting the translocation of DNA through a nanopore using graphene nanoribbons. Nat. Nanotechnol. 2013, 8, 939–945. [Google Scholar] [CrossRef]
- Riedl, J.; Ding, Y.; Fleming, A.M.; Burrows, C.J. Identification of DNA lesions using a third base pair for amplification and nanopore sequencing. Nat. Commun. 2015, 6, 8807. [Google Scholar] [CrossRef]
- Fleming, A.M.; Mathewson, N.J.; Howpay Manage, S.A.; Burrows, C.J. Nanopore Dwell Time Analysis Permits Sequencing and Conformational Assignment of Pseudouridine in SARS-CoV-2. ACS Cent. Sci. 2021, 7, 1707–1717. [Google Scholar] [CrossRef]
- Yan, S.; Wang, L.; Wang, Y.; Cao, Z.; Zhang, S.; Du, X.; Fan, P.; Zhang, P.; Chen, H.Y.; Huang, S. Non-binary Encoded Nucleic Acid Barcodes Directly Readable by a Nanopore. Angew. Chem. Int. Ed. 2022, 61, e202116482. [Google Scholar] [CrossRef]
- Jia, W.; Hu, C.; Wang, Y.; Liu, Y.; Wang, L.; Zhang, S.; Zhu, Q.; Gu, Y.; Zhang, P.; Ma, J.; et al. Identification of Single-Molecule Catecholamine Enantiomers Using a Programmable Nanopore. ACS Nano 2022, 16, 6615–6624. [Google Scholar] [CrossRef]
- Liu, P.; Kawano, R. Recognition of Single-Point Mutation Using a Biological Nanopore. Small Methods 2020, 4, 2000101. [Google Scholar] [CrossRef]
- Wang, F.; Zahid, O.K.; Swain, B.E.; Parsonage, D.; Hollis, T.; Harvey, S.; Perrino, F.W.; Kohli, R.M.; Taylor, E.W.; Hall, A.R. Solid-State Nanopore Analysis of Diverse DNA Base Modifications Using a Modular Enzymatic Labeling Process. Nano Lett. 2017, 17, 7110–7116. [Google Scholar] [CrossRef] [PubMed]
- Ying, Y.L.; Zhang, J.; Gao, R.; Long, Y.T. Nanopore-based sequencing and detection of nucleic acids. Angew Chem. Int. Ed. Engl. 2013, 52, 13154–13161. [Google Scholar] [CrossRef] [PubMed]
- Wang, L.; Chen, X.; Zhou, S.; Roozbahani, G.M.; Zhang, Y.; Wang, D.; Guan, X. Displacement chemistry-based nanopore analysis of nucleic acids in complicated matrices. ChemComm 2018, 54, 13977–13980. [Google Scholar] [CrossRef] [PubMed]
- Workman, R.E.; Tang, A.D.; Tang, P.S.; Jain, M.; Tyson, J.R.; Razaghi, R.; Zuzarte, P.C.; Gilpatrick, T.; Payne, A.; Quick, J.; et al. Nanopore native RNA sequencing of a human poly(A) transcriptome. Nat. Methods 2019, 16, 1297–1305. [Google Scholar] [CrossRef]
- Nakane, J.J.; Akeson, M.; Marziali, A. Nanopore sensors for nucleic acid analysis. J. Phys. Condens. Matter 2003, 15, R1365. [Google Scholar] [CrossRef]
- Deamer, D.W.; Branton, D. Characterization of nucleic acids by nanopore analysis. Acc. Chem. Res. 2002, 35, 817–825. [Google Scholar] [CrossRef]
- Derrington, I.M.; Craig, J.M.; Stava, E.; Laszlo, A.H.; Ross, B.C.; Brinkerhoff, H.; Nova, I.C.; Doering, K.; Tickman, B.I.; Ronaghi, M.; et al. Subangstrom single-molecule measurements of motor proteins using a nanopore. Nat. Biotechnol. 2015, 33, 1073–1075. [Google Scholar] [CrossRef]
- Yusko, E.C.; Bruhn, B.R.; Eggenberger, O.M.; Houghtaling, J.; Rollings, R.C.; Walsh, N.C.; Nandivada, S.; Pindrus, M.; Hall, A.R.; Sept, D.; et al. Real-time shape approximation and fingerprinting of single proteins using a nanopore. Nat. Nanotechnol. 2017, 12, 360–367. [Google Scholar] [CrossRef]
- Waduge, P.; Hu, R.; Bandarkar, P.; Yamazaki, H.; Cressiot, B.; Zhao, Q.; Whitford, P.C.; Wanunu, M. Nanopore-Based Measurements of Protein Size, Fluctuations, and Conformational Changes. ACS Nano 2017, 11, 5706–5716. [Google Scholar] [CrossRef]
- Wei, X.; Ma, D.; Zhang, Z.; Wang, L.Y.; Gray, J.L.; Zhang, L.; Zhu, T.; Wang, X.; Lenhart, B.J.; Yin, Y.; et al. N-Terminal Derivatization-Assisted Identification of Individual Amino Acids Using a Biological Nanopore Sensor. ACS Sens. 2020, 5, 1707–1716. [Google Scholar] [CrossRef] [PubMed]
- Wloka, C.; Van Meervelt, V.; van Gelder, D.; Danda, N.; Jager, N.; Williams, C.P.; Maglia, G. Label-Free and Real-Time Detection of Protein Ubiquitination with a Biological Nanopore. ACS Nano 2017, 11, 4387–4394. [Google Scholar] [CrossRef]
- Afshar Bakshloo, M.; Kasianowicz, J.J.; Pastoriza-Gallego, M.; Mathé, J.; Daniel, R.; Piguet, F.; Oukhaled, A. Nanopore-Based Protein Identification. J. Am. Chem. Soc. 2022, 144, 2716–2725. [Google Scholar] [CrossRef] [PubMed]
- Shimizu, K.; Mijiddorj, B.; Usami, M.; Mizoguchi, I.; Yoshida, S.; Akayama, S.; Hamada, Y.; Ohyama, A.; Usui, K.; Kawamura, I.; et al. De novo design of a nanopore for single-molecule detection that incorporates a β-hairpin peptide. Nat. Nanotechnol. 2022, 17, 67–75. [Google Scholar] [CrossRef] [PubMed]
- Zhou, S.; Wang, H.; Chen, X.; Wang, Y.; Zhou, D.; Liang, L.; Wang, L.; Wang, D.; Guan, X. Single-molecule Study on the Interactions between Cyclic Nonribosomal Peptides and Protein Nanopore. ACS Appl. Bio Mater. 2020, 3, 554–560. [Google Scholar] [CrossRef] [PubMed]
- Reiner, J.E.; Kasianowicz, J.J.; Nablo, B.J.; Robertson, J.W. Theory for polymer analysis using nanopore-based single-molecule mass spectrometry. Proc. Natl. Acad. Sci. USA 2010, 107, 12080–12085. [Google Scholar] [CrossRef]
- Neher, E.; Sakmann, B. Single-channel currents recorded from membrane of denervated frog muscle fibres. Nature 1976, 260, 799–802. [Google Scholar] [CrossRef]
- Ishii, Y.; Yanagida, T. Single molecule detection in life sciences. Single Mol. 2000, 1, 5–16. [Google Scholar] [CrossRef]
- Arroyo, J.O.; Kukura, P. Non-fluorescent schemes for single-molecule detection, imaging and spectroscopy. Nat. Photonics 2016, 10, 11–17. [Google Scholar] [CrossRef]
- Lelek, M.; Gyparaki, M.T.; Beliu, G.; Schueder, F.; Griffié, J.; Manley, S.; Jungmann, R.; Sauer, M.; Lakadamyali, M.; Zimmer, C. Single-molecule localization microscopy. Nat. Rev. Methods Primers 2021, 1, 39. [Google Scholar] [CrossRef]
- Yuana, Y.; Oosterkamp, T.H.; Bahatyrova, S.; Ashcroft, B.; Garcia Rodriguez, P.; Bertina, R.M.; Osanto, S. Atomic force microscopy: A novel approach to the detection of nanosized blood microparticles. J. Thromb. Haemost. 2010, 8, 315–323. [Google Scholar] [CrossRef] [PubMed]
- Li, Y.; Zhao, L.; Yao, Y.; Guo, X. Single-Molecule Nanotechnologies: An Evolution in Biological Dynamics Detection. ACS Appl. Bio Mater. 2020, 3, 68–85. [Google Scholar] [CrossRef]
- Jain, M.; Olsen, H.E.; Paten, B.; Akeson, M. The Oxford Nanopore MinION: Delivery of nanopore sequencing to the genomics community. Genome Biol. 2016, 17, 239. [Google Scholar] [CrossRef]
- Huo, W.; Ling, W.; Wang, Z.; Ya, L.; Zhou, M.; Ren, M.; Li, X.; Li, J.; Xia, Z.; Liu, X.; et al. Miniaturized DNA Sequencers for Personal Use: Unreachable Dreams or Achievable Goals. Front. Nanotechnol. 2021, 3, 628861. [Google Scholar] [CrossRef]
- Maglia, G.; Heron, A.J.; Stoddart, D.; Japrung, D.; Bayley, H. Analysis of single nucleic acid molecules with protein nanopores. Meth. Enzymol. 2010, 475, 591–623. [Google Scholar]
- Deamer, D.; Akeson, M.; Branton, D. Three decades of nanopore sequencing. Nat. Biotechnol. 2016, 34, 518–524. [Google Scholar] [CrossRef] [PubMed]
- Firnkes, M.; Pedone, D.; Knezevic, J.; Döblinger, M.; Rant, U. Electrically facilitated translocations of proteins through silicon nitride nanopores: Conjoint and competitive action of diffusion, electrophoresis, and electroosmosis. Nano Lett. 2010, 10, 2162–2167. [Google Scholar] [CrossRef] [PubMed]
- Song, L.; Hobaugh, M.R.; Shustak, C.; Cheley, S.; Bayley, H.; Gouaux, J.E. Structure of staphylococcal alpha-hemolysin, a heptameric transmembrane pore. Science 1996, 274, 1859–1866. [Google Scholar] [CrossRef]
- Walker, B.W.; Kasianowicz, J.J.; Krishnasastry, M.V.; Bayley, H. A pore-forming protein with a metal-actuated switch. Prod. Eng. 1994, 7, 655–662. [Google Scholar] [CrossRef]
- Kasianowicz, J.J.; Brandin, E.; Branton, D.; Deamer, D. Characterization of Individual Polynucleotide Molecules Using a Membrane Channel. Proc. Natl. Acad. Sci. USA 1996, 93, 13770–13773. [Google Scholar] [CrossRef]
- Akeson, M.; Branton, D.; Kasianowicz, J.J.; Brandin, E.; Deamer, D.W. Microsecond time-scale discrimination among polycytidylic acid, polyadenylic acid, and polyuridylic acid as homopolymers or as segments within single RNA molecules. Biophys. J. 1999, 77, 3227–3233. [Google Scholar] [CrossRef]
- Butler, T.Z.; Pavlenok, M.; Derrington, I.M.; Niederweis, M.; Gundlach, J.H. Single-molecule DNA detection with an engineered MspA protein nanopore. Proc. Natl. Acad. Sci. USA 2008, 105, 20647–20652. [Google Scholar] [CrossRef]
- Manrao, E.A.; Derrington, I.M.; Laszlo, A.H.; Langford, K.W.; Hopper, M.K.; Gillgren, N.; Pavlenok, M.; Niederweis, M.; Gundlach, J.H. Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase. Nat. Biotechnol. 2012, 30, 349–353. [Google Scholar] [CrossRef]
- Manrao, E.A.; Derrington, I.M.; Pavlenok, M.; Niederweis, M.; Gundlach, J.H. Nucleotide discrimination with DNA immobilized in the MspA nanopore. PLoS ONE 2011, 6, e25723. [Google Scholar] [CrossRef] [PubMed]
- Wang, Y.; Guan, X.; Zhang, S.; Liu, Y.; Wang, S.; Fan, P.; Du, X.; Yan, S.; Zhang, P.; Chen, H.-Y. Structural-profiling of low molecular weight RNAs by nanopore trapping/translocation using Mycobacterium smegmatis porin A. Nat. Commun. 2021, 12, 1–14. [Google Scholar] [CrossRef] [PubMed]
- Roozbahani, G.M.; Chen, X.; Zhang, Y.; Wang, L.; Guan, X. Nanopore detection of metal ions: Current status and future directions. Small Methods 2020, 4, 2000266. [Google Scholar] [CrossRef] [PubMed]
- Soskine, M.; Biesemans, A.; Moeyaert, B.; Cheley, S.; Bayley, H.; Maglia, G. An engineered ClyA nanopore detects folded target proteins by selective external association and pore entry. Nano Lett. 2012, 12, 4895–4900. [Google Scholar] [CrossRef]
- Wang, Y.; Gu, L.Q.; Tian, K. The aerolysin nanopore: From peptidomic to genomic applications. Nanoscale 2018, 10, 13857–13866. [Google Scholar] [CrossRef]
- Long, Z.; Zhan, S.; Gao, P.; Wang, Y.; Lou, X.; Xia, F. Recent Advances in Solid Nanopore/Channel Analysis. Anal. Chem. 2018, 90, 577–588. [Google Scholar] [CrossRef]
- Howorka, S.; Siwy, Z. Nanopore analytics: Sensing of single molecules. Chem. Soc. Rev. 2009, 38, 2360–2384. [Google Scholar] [CrossRef]
- van den Berg, A.; Wessling, M. Silicon for the perfect membrane. Nature 2007, 445, 726. [Google Scholar] [CrossRef] [PubMed]
- Hu, R.; Tong, X.; Zhao, Q. Four Aspects about Solid-State Nanopores for Protein Sensing: Fabrication, Sensitivity, Selectivity, and Durability. Adv. Healthc. Mater. 2020, 9, 2000933. [Google Scholar] [CrossRef] [PubMed]
- Kleefen, A.; Pedone, D.; Grunwald, C.; Wei, R.; Firnkes, M.; Abstreiter, G.; Rant, U.; Tampé, R. Multiplexed Parallel Single Transport Recordings on Nanopore Arrays. Nano Lett. 2010, 10, 5080–5087. [Google Scholar] [CrossRef] [PubMed]
- Roman, J.; Jarroux, N.; Patriarche, G.; Français, O.; Pelta, J.; Le Pioufle, B.; Bacri, L. Functionalized Solid-State Nanopore Integrated in a Reusable Microfluidic Device for a Better Stability and Nanoparticle Detection. ACS Appl. Mater. Interfaces 2017, 9, 41634–41640. [Google Scholar] [CrossRef] [PubMed]
- Storm, A.J.; Chen, J.H.; Ling, X.S.; Zandbergen, H.W.; Dekker, C. Fabrication of solid-state nanopores with single-nanometre precision. Nat. Mater. 2003, 2, 537–540. [Google Scholar] [CrossRef] [PubMed]
- Freedman, K.J.; Otto, L.M.; Ivanov, A.P.; Barik, A.; Oh, S.-H.; Edel, J.B. Nanopore sensing at ultra-low concentrations using single-molecule dielectrophoretic trapping. Nat. Commun. 2016, 7, 10217. [Google Scholar] [CrossRef]
- Garaj, S.; Hubbard, W.; Reina, A.; Kong, J.; Branton, D.; Golovchenko, J.A. Graphene as a subnanometre trans-electrode membrane. Nature 2010, 467, 190–193. [Google Scholar] [CrossRef]
- Gasparyan, L.; Mazo, I.; Simonyan, V.; Gasparyan, F. DNA Sequencing: Current State and Prospects of Development. Biophys. J. 2019, 09, 169–197. [Google Scholar] [CrossRef]
- Shi, W.; Friedman, A.K.; Baker, L.A. Nanopore Sensing. Anal. Chem. 2017, 89, 157–188. [Google Scholar] [CrossRef]
- Schibel, A.E.; An, N.; Jin, Q.; Fleming, A.M.; Burrows, C.J.; White, H.S. Nanopore detection of 8-oxo-7,8-dihydro-2′-deoxyguanosine in immobilized single-stranded DNA via adduct formation to the DNA damage site. J. Am. Chem. Soc. 2010, 132, 17992–17995. [Google Scholar] [CrossRef]
- Perera, R.T.; Fleming, A.M.; Johnson, R.P.; Burrows, C.J.; White, H.S. Detection of benzo[a]pyrene-guanine adducts in single-stranded DNA using the α-hemolysin nanopore. Nanotechnology 2015, 26, 074002. [Google Scholar] [CrossRef] [PubMed]
- Rouse, J.; Jackson, S.P. Interfaces between the detection, signaling, and repair of DNA damage. Science 2002, 297, 547–551. [Google Scholar] [CrossRef] [PubMed]
- Zhou, B.B.; Elledge, S.J. The DNA damage response: Putting checkpoints in perspective. Nature 2000, 408, 433–439. [Google Scholar] [CrossRef] [PubMed]
- Ma, F.; Yan, S.; Zhang, J.; Wang, Y.; Wang, L.; Wang, Y.; Zhang, S.; Du, X.; Zhang, P.; Chen, H.-Y.; et al. Nanopore Sequencing Accurately Identifies the Cisplatin Adduct on DNA. ACS Sens. 2021, 6, 3082–3092. [Google Scholar] [CrossRef] [PubMed]
- Zhang, J.; Wang, Y.; Wang, Y.; Zhang, P.; Chen, H.-Y.; Huang, S. Discrimination between Different DNA Lesions by Monitoring Single-Molecule Polymerase Stalling Kinetics during Nanopore Sequencing. Nano Lett. 2022, 22, 5561–5569. [Google Scholar] [CrossRef]
- Taylor, A.I.; Arangundy-Franklin, S.; Holliger, P. Towards applications of synthetic genetic polymers in diagnosis and therapy. Curr. Opin. Chem. Biol. 2014, 22, 79–84. [Google Scholar] [CrossRef]
- Feldman, A.W.; Romesberg, F.E. Expansion of the Genetic Alphabet: A Chemist’s Approach to Synthetic Biology. Acc. Chem. Res. 2018, 51, 394–403. [Google Scholar] [CrossRef]
- Pinheiro, V.B.; Arangundy-Franklin, S.; Holliger, P. Compartmentalized Self-Tagging for In Vitro-Directed Evolution of XNA Polymerases. Curr. Protoc. Nucleic Acid Chem. 2014, 57, 9. [Google Scholar] [CrossRef]
- Yan, S.; Li, X.; Zhang, P.; Wang, Y.; Chen, H.Y.; Huang, S.; Yu, H. Direct sequencing of 2′-deoxy-2′-fluoroarabinonucleic acid (FANA) using nanopore-induced phase-shift sequencing (NIPSS). Chem. Sci. 2019, 10, 3110–3117. [Google Scholar] [CrossRef]
- Piguet, F.; Ouldali, H.; Pastoriza-Gallego, M.; Manivet, P.; Pelta, J.; Oukhaled, A. Identification of single amino acid differences in uniformly charged homopolymeric peptides with aerolysin nanopore. Nat. Commun. 2018, 9, 966. [Google Scholar] [CrossRef]
- Movileanu, L.; Schmittschmitt, J.P.; Scholtz, J.M.; Bayley, H. Interactions of peptides with a protein pore. Biophys. J. 2005, 89, 1030–1045. [Google Scholar] [CrossRef] [PubMed]
- Yan, S.; Zhang, J.; Wang, Y.; Guo, W.; Zhang, S.; Liu, Y.; Cao, J.; Wang, Y.; Wang, L.; Ma, F.; et al. Single Molecule Ratcheting Motion of Peptides in a Mycobacterium smegmatis Porin A (MspA) Nanopore. Nano Lett. 2021, 21, 6703–6710. [Google Scholar] [CrossRef] [PubMed]
- Brinkerhoff, H.; Kang, A.S.; Liu, J.; Aksimentiev, A.; Dekker, C. Multiple rereads of single proteins at single–amino acid resolution using nanopores. Science 2021, 374, 1509–1513. [Google Scholar] [CrossRef] [PubMed]
- Jia, W.; Hu, C.; Wang, Y.; Gu, Y.; Qian, G.; Du, X.; Wang, L.; Liu, Y.; Cao, J.; Zhang, S.; et al. Programmable nano-reactors for stochastic sensing. Nat. Commun. 2021, 12, 5811. [Google Scholar] [CrossRef] [PubMed]
- Guo, J.; Amini, S.; Lei, Q.; Ping, Y.; Agola, J.O.; Wang, L.; Zhou, L.; Cao, J.; Franco, S.; Noureddine, A.; et al. Robust and Long-Term Cellular Protein and Enzymatic Activity Preservation in Biomineralized Mammalian Cells. ACS Nano 2022, 16, 2164–2175. [Google Scholar] [CrossRef] [PubMed]
- Baoutina, A.; Bhat, S.; Partis, L.; Emslie, K.R. Storage Stability of Solutions of DNA Standards. Anal. Chem. 2019, 91, 12268–12274. [Google Scholar] [CrossRef] [PubMed]
- Shendure, J.; Balasubramanian, S.; Church, G.M.; Gilbert, W.; Rogers, J.; Schloss, J.A.; Waterston, R.H. DNA sequencing at 40: Past, present and future. Nature 2017, 550, 345–353. [Google Scholar] [CrossRef]
- Kohll, A.X.; Antkowiak, P.L.; Chen, W.D.; Nguyen, B.H.; Stark, W.J.; Ceze, L.; Strauss, K.; Grass, R.N. Stabilizing synthetic DNA for long-term data storage with earth alkaline salts. ChemComm 2020, 56, 3613–3616. [Google Scholar] [CrossRef]
- Hao, Y.; Li, Q.; Fan, C.; Wang, F. Data Storage Based on DNA. Small Struct. 2021, 2, 2000046. [Google Scholar] [CrossRef]
- Lopez, R.; Chen, Y.-J.; Dumas Ang, S.; Yekhanin, S.; Makarychev, K.; Racz, M.Z.; Seelig, G.; Strauss, K.; Ceze, L. DNA assembly for nanopore data storage readout. Nat. Commun. 2019, 10, 2933. [Google Scholar] [CrossRef]
- Liu, H.; Wang, J.; Song, S.; Fan, C.; Gothelf, K.V. A DNA-based system for selecting and displaying the combined result of two input variables. Nat. Commun. 2015, 6, 10089. [Google Scholar] [CrossRef] [PubMed]
- Ge, Z.; Gu, H.; Li, Q.; Fan, C. Concept and Development of Framework Nucleic Acids. J. Am. Chem. Soc. 2018, 140, 17808–17819. [Google Scholar] [CrossRef] [PubMed]
- Chen, K.; Kong, J.; Zhu, J.; Ermann, N.; Predki, P.; Keyser, U.F. Digital Data Storage Using DNA Nanostructures and Solid-State Nanopores. Nano Lett. 2019, 19, 1210–1215. [Google Scholar] [CrossRef] [PubMed]
- Bell, N.A.W.; Keyser, U.F. Digitally encoded DNA nanostructures for multiplexed, single-molecule protein sensing with nanopores. Nat. Nanotechnol. 2016, 11, 645–651. [Google Scholar] [CrossRef]
- Cao, C.; Krapp, L.F.; Al Ouahabi, A.; König, N.F.; Cirauqui, N.; Radenovic, A.; Lutz, J.F.; Peraro, M.D. Aerolysin nanopores decode digital information stored in tailored macromolecular analytes. Sci. Adv. 2020, 6, eabc2661. [Google Scholar] [CrossRef]
- LeCun, Y.; Boser, B.E.; Denker, J.S.; Henderson, D.; Howard, R.E.; Hubbard, W.E.; Jackel, L.D. Backpropagation Applied to Handwritten Zip Code Recognition. Neural Comput 1989, 1, 541–551. [Google Scholar] [CrossRef]
- Zaremba, W.; Sutskever, I.; Vinyals, O. Recurrent Neural Network Regularization. arXiv 2014, arXiv:1409.2329. [Google Scholar]
- Hinton, G.E.; Salakhutdinov, R.R. Reducing the dimensionality of data with neural networks. Science 2006, 313, 504–507. [Google Scholar] [CrossRef]
- Taniguchi, M.; Minami, S.; Ono, C.; Hamajima, R.; Morimura, A.; Hamaguchi, S.; Akeda, Y.; Kanai, Y.; Kobayashi, T.; Kamitani, W.; et al. Combining machine learning and nanopore construction creates an artificial intelligence nanopore for coronavirus detection. Nat. Commun. 2021, 12, 3726. [Google Scholar] [CrossRef]
- Arima, A.; Tsutsui, M.; Washio, T.; Baba, Y.; Kawai, T. Solid-state nanopore platform integrated with machine learning for digital diagnosis of virus infection. Anal. Chem. 2020, 93, 215–227. [Google Scholar] [CrossRef]
- Ledergerber, C.; Dessimoz, C. Base-calling for next-generation sequencing platforms. Brief. Bioinform. 2011, 12, 489–497. [Google Scholar] [CrossRef] [PubMed]
- David, M.; Dursi, L.J.; Yao, D.; Boutros, P.C.; Simpson, J.T. Nanocall: An open source basecaller for Oxford Nanopore sequencing data. Bioinformatics 2017, 33, 49–55. [Google Scholar] [CrossRef]
- Bo?a, V.; Brejová, B.; Vina?, T. DeepNano: Deep recurrent neural networks for base calling in MinION nanopore reads. PLoS ONE 2017, 12, e0178751. [Google Scholar] [CrossRef] [PubMed]
- Stoiber, M.; Brown, J. BasecRAWller: Streaming Nanopore Basecalling Directly from Raw Signal. bioRxiv 2017, 133058. [Google Scholar] [CrossRef]
- Teng, H.; Cao, M.D.; Hall, M.B.; Duarte, T.; Wang, S.; Coin, L.J.M. Chiron: Translating nanopore raw signal directly into nucleotide sequence using deep learning. GigaScience 2018, 7, giy037. [Google Scholar] [CrossRef]
- Liu, Q.; Fang, L.; Yu, G.; Wang, D.; Xiao, C.-L.; Wang, K. Detection of DNA base modifications by deep recurrent neural network on Oxford Nanopore sequencing data. Nat. Commun. 2019, 10, 2449. [Google Scholar] [CrossRef]
- Ni, P.; Huang, N.; Nie, F.; Zhang, J.; Zhang, Z.; Wu, B.; Bai, L.; Liu, W.; Xiao, C.-L.; Luo, F.; et al. Genome-wide detection of cytosine methylations in plant from Nanopore data using deep learning. Nat. Commun. 2021, 12, 5976. [Google Scholar] [CrossRef]
- Ni, P.; Huang, N.; Zhang, Z.; Wang, D.P.; Liang, F.; Miao, Y.; Xiao, C.L.; Luo, F.; Wang, J. DeepSignal: Detecting DNA methylation state from Nanopore sequencing reads using deep-learning. Bioinformatics 2019, 35, 4586–4595. [Google Scholar] [CrossRef]
- Doroschak, K.; Zhang, K.; Queen, M.; Mandyam, A.; Strauss, K.; Ceze, L.; Nivala, J. Rapid and robust assembly and decoding of molecular tags with DNA-based nanopore signatures. Nat. Commun. 2020, 11, 5454. [Google Scholar] [CrossRef]
- Buterez, D. Scaling up DNA digital data storage by efficiently predicting DNA hybridisation using deep learning. Sci. Rep. 2021, 11, 20517. [Google Scholar] [CrossRef]
- Rang, F.J.; Kloosterman, W.P.; de Ridder, J. From squiggle to basepair: Computational approaches for improving nanopore sequencing read accuracy. Genome Biol. 2018, 19, 90. [Google Scholar] [CrossRef] [PubMed]
- Bayley, H. Nanopore sequencing: From imagination to reality. Clin. Chem. 2015, 61, 25–31. [Google Scholar] [CrossRef] [PubMed]
声明: 此文观点不代表本站立场;转载须要保留原文链接;版权疑问请联系我们。